インパクトファクターの高いものを選んでもらえるかな?
医学論文にはインパクトファクターといって、雑誌の引用数などによってきまるそのジャーナルの権威性があります。
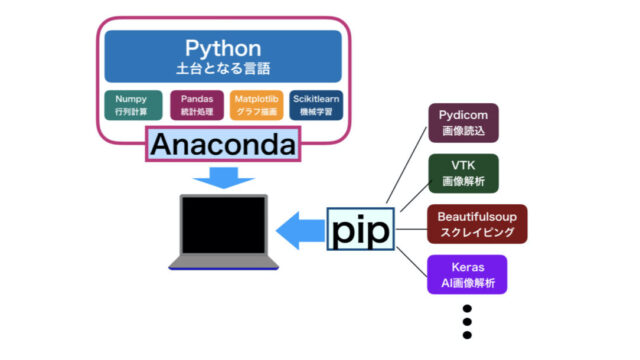
今回は複数の論文(大体500個くらいの論文)からインパクトファクターを調べる機会があり、Pythonのプログラミングをフル活用することでうまくいったので、方法を解説します。
- Google Chromeの「Pubmed Impact Factor」を使用
- Seleniumで目的のidの論文を開く
- BeautifulSoupでインパクトファクターを入手
ちょっと力技なのですが、
ひとつずつ解説します。
①Google Chromeのプラグイン「Pubmed Impact Factor」
Google Chromeで「Pubmed Impact Factor」といれると、その名の拡張機能が出てきます。
この拡張機能をインストールすると、Pubmedを開いた時、
その雑誌のインパクトファクターが表示されるようになります。

↓ 拡張機能インストール後

普段のpubmedでは自分が知らない雑誌のインパクトファクターはわからないので、これはとても便利な機能です。
さらに、設定で
「一定のインパクトファクターより低いものは表示しない」
というオプションを設定することができます。

この機能をあらかじめGoogle Chromeにインストールしておき、
目的の論文が開いたときに、インパクトファクターが表示してあるようにしておきます。
② Seleniumを利用して論文を自動で表示させる
前提として私は
Selenium 4
を使用しています。
1つ前のバージョンの Selenium 3だと
設定が微妙に変わってくるので注意が必要です。
インストールしていない場合は、ターミナルまたはコマンドプロンプトから
pip install selenium
でインストールできます。
(1)WebdriverでChromeを起動させ、Pubmed Impact factorをインストール
PythonのインタープリターからSeleniumのWebdriverを起動させます。
from selenium import webdriver
from selenium.webdriver.common.by import By
driver = webdriver.Chrome()pythonのwebdriverから動かせるChromeを立ち上げます。
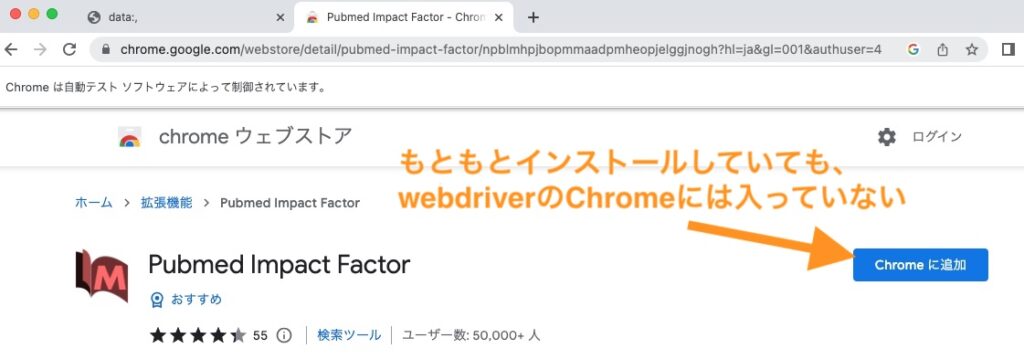
ここでちょっと厄介なのですが、
このChromeは普段つかっているChromeと異なるため、
このChromeの画面を操作して拡張機能
「Pubmed Impact Factor」をインストールします。
そうすると、こちらのChromeでもPubmedの画面にインパクトファクターが表示されるようになります。
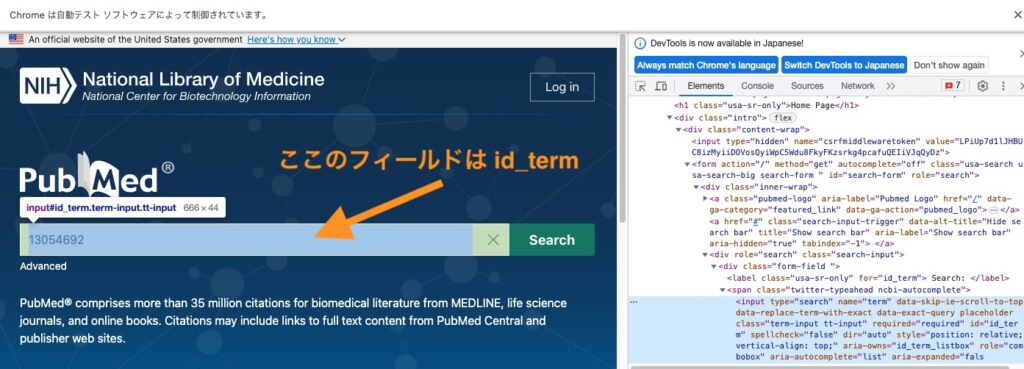
(2) Pubmedのページを表示し、PMIDから目的の論文のページを開く
以下のスクリプトを入力して、目的のidの論文のページを開きます。
pmid = 13054692 #有名なDNAの論文をidに 設定
"""Pubmedのホームを開く"""
driver.get('https://pubmed.ncbi.nlm.nih.gov')
"""id_termのフィールドにpmid(論文id)を入力する"""
search_term = driver.find_element(By.ID,"id_term")
search_term.send_keys(pmid)
"""「Search」ボタンをクリックする"""
search_button = driver.find_element(By.CLASS_NAME, 'search-btn')
search_button.click()
設定
"""Pubmedのホームを開く"""
driver.get('https://pubmed.ncbi.nlm.nih.gov')
"""id_termのフィールドにpmid(論文id)を入力する"""
search_term = driver.find_element(By.ID,"id_term")
search_term.send_keys(pmid)
"""「Search」ボタンをクリックする"""
search_button = driver.find_element(By.CLASS_NAME, 'search-btn')
search_button.click()これで入力したpmidのページが開きます。

自動で入力、クリックされ、目的のページが開く。
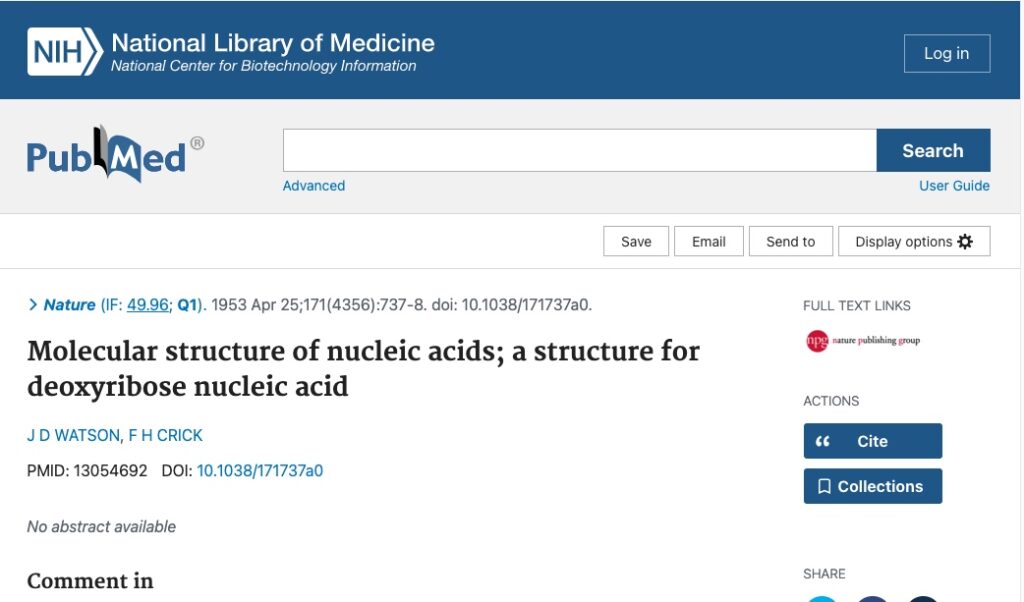
③ BeautifulSoupでインパクトファクターを取得
開いたページにはインパクトファクターが記載されているので、この数字を入手します。
from bs4 import BeautifulSoup
"""driverで開いているページをBeautifulSoupにわたす"""
soup = BeautifulSoup(driver.page_source, 'html.parser')
"""class「if」という名前のspanフィールドのテキスト情報を入手"""
impact_factor = soup.find('span', class_='if').textwebdriverから表示されているページの情報をBeautifulSoupにわたし、classがif(インパクトファクター)のspanタグをとりだすと、インパクトファクターがわかります。
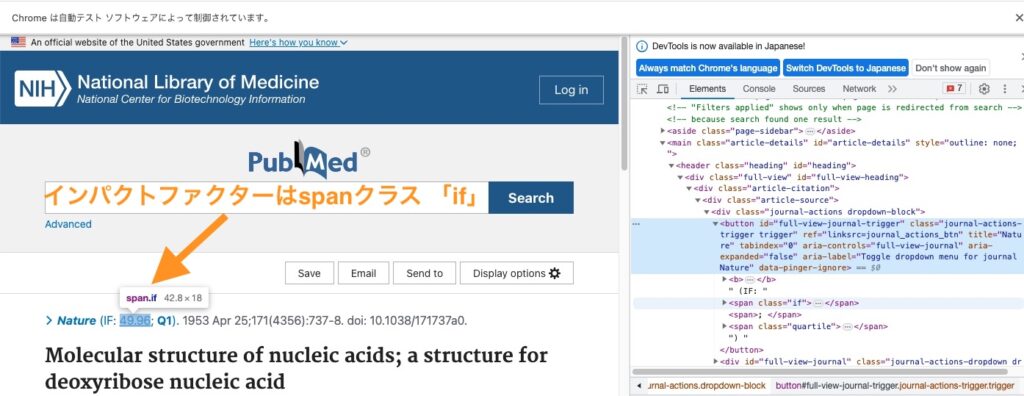
print(impact_factor)49.96
これをすべてのPMIDのリストについてForなどを用いて繰り返し行えば、
それぞれのインパクトファクターが出てくるわけです。
Pubmedの抄録自動でChatGPTに翻訳させるプログラムも紹介していきますね。。