Pubmed
https://pubmed.ncbi.nlm.nih.gov
は世界的に最も有名な医学論文の検索サイトです。
このページではPythonの簡単なプログラミングを使ってPubmedからデータを取得する方法を解説します。
目標としては、
検索語から論文のID、pmidをリストで作成してくれる
search_articles
というプログラムと
論文IDを入力すると、文献情報、ここではアブストラクト(抄録)を返してくれる
fetch_abstract
という2つのプログラムを作成します。
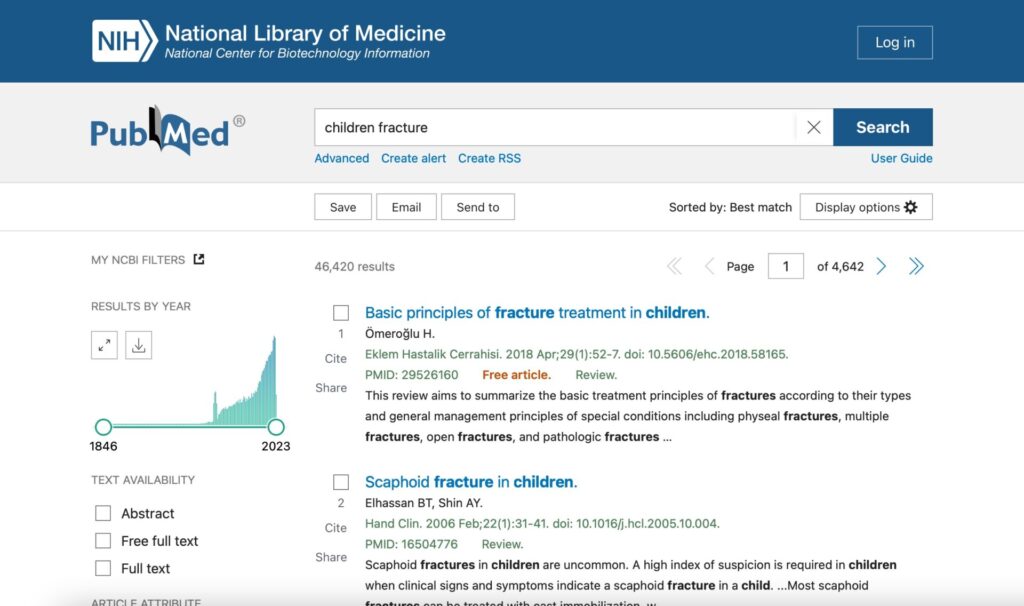
例えば、このPubmedのページで小児の骨折、
“children fracture”を入力すると
以下のように表示されます。
この検索機能をpythonから使うことができます。先程の関数を定義して使用することによって
results = search_articles(‘children fracture’)
print(results[‘IdList’])
[‘29526160’, ‘16504776’, ‘32868623’, ‘15652934’, ‘35089608’, ‘2880651’, ‘34282089’, ‘14623836’, ‘29369266’, ‘23545729’・・・
と検索結果の論文のIDをリストとして取得でき、
一番はじめの論文のidの論文からアブストラクトを抽出すると
fetch_abstract(29526160)
‘This review aims to summarize the basic treatment principles of fractures according to their types and general management principles of special conditions including physeal fractures・・・
とアブストラクトを表示することができます。
これは検索サイトで表示される結果と一致しています。
この方法ができると、
Pubmedのホームページを開かなくても、
好きな検索ワードで、好きな文献のアブストラクト、タイトルや、publicationtype、出版年といった情報を一気に入手し、
pythonで操作することができます。
このような便利な関数を作成する流れと、その内容を説明していきます。
1.Biopythonをインストールする
まず、ターミナル、またはコマンドプロンプトで、pipを使用してBiopythonをインストールしておきます。
pip install biopython
Biopythonは、生物学やバイオインフォマティクスの研究者けのPythonツールボックスです。生物学的なデータの処理、解析、可視化をサポートするために開発されており、多くの機能が備わっています。
その一つに
「Pubmedから情報を取得する」
という
「Entrez」
というモジュール機能(ツール)があります。
今回はその機能を使用します。
2.検索キーワードから論文IDリストを取得する
次にPythonのスクリプトを作成します
プログラムを作成するにあたり、使う人のemailが必要になります。
from Bio import Entrez
Entrez.email = "使う人のEmailアドレス"
def search_articles(keyword, max_results=10):
handle = Entrez.esearch(db='pubmed',
sort='relevance',
retmax=str(max_results),
term=keyword)
results = Entrez.read(handle)
return results
が検索を行う関数のスクリプトになります。
Emailアドレスには使用する人のアドレスを入力する必要があります。
Entrez.esearch()
というのは、Biopythonのツールで、Pubmedにアクセスして結果を返してくれます。
Entrez.esearch(db, sort, retmax, term)
db:どのデータベースで検索するか。ここでは「pubmed」に設定
sort:並び順、ここでは’relevance’関連性を指定
retmax:取得する最大結果数
term:検索キーワード
handleは検索を入力するとホームページ上で帰ってきたレスポンスで、これを
Entrez.read()関数でPythonの辞書の形に変換したのが
resultsに入ります。
print(results)とすると、
print(results)
{'Count': '15759',
'RetMax': '10',
'RetStart': '0',
'IdList': ['34479569', '27921119', '30983255', '32980938', '31965305', '31180787', '29205983', '36345194', '29499821', '26808044'],
'TranslationSet': [{'From': 'humerus, fracture', 'To': '"humeral fractures"[MeSH Terms] OR ("humeral"[All Fields] AND "fractures"[All Fields]) OR "humeral fractures"[All Fields] OR ("humerus"[All Fields] AND "fracture"[All Fields]) OR "humerus fracture"[All Fields]'}], 'QueryTranslation': '"humeral fractures"[MeSH Terms] OR ("humeral"[All Fields] AND "fractures"[All Fields]) OR "humeral fractures"[All Fields] OR ("humerus"[All Fields] AND "fracture"[All Fields]) OR "humerus fracture"[All Fields]'}と、えらい長い構造になっています。
このIdListが文献のidのリストです。(ここではpubmedなのでpmid)
ちなみに、’TranslationSet’という中をみると、実際に入力した言葉が、よりあいまいさを減らすため、なかでどのような検索条件に変更されているか、ということがわかります。
pubmedのアドバンスモードを使用した人ならわかりやすいかもしれません。
3.Pubmedのid(PMID)から論文の情報を取得する
つぎに、pubmedのid, 「PMID」からアブストラクトを含めた論文の詳細を入手する関数を作成します。
関数の名前は
「fetch_abstract」
とします。
def fetch_abstract(pmid):
handle = Entrez.efetch(db='pubmed', id=pmid, retmode='xml')
article = Entrez.read(handle)['PubmedArticle'][0]
abstract = article['MedlineCitation']['Article']['Abstract']['AbstractText'][0]
return abstract
が作成する関数のスクリプトです。
ここで、
Entrez.efetch()という関数は、以下を設定して、論文の情報を取得します。
Entrez.efetch(db, id, retmode)
db:どのデータベースで検索するか
id:論文のID。ここではpmidを入力
retmode:返す形式、xmlという形式に指定
ここでは例として、ノーベル賞を受賞した整形外科医、山中先生の
iPS細胞の論文 PMID 16904174
を参照していきます。
Entrez.read()関数で
ホームページから入手したxml形式の情報をpythonの情報に
変換します。
入手した情報を表示してみると
handle = Entrez.efetch(db='pubmed', id=16904174, retmode='xml')
article = Entrez.read(handle)['PubmedArticle'][0]
print(article)
{‘PubmedArticle’:
[{‘MedlineCitation’: DictElement(
{‘OtherID’: [],
‘OtherAbstract’: [],
‘SpaceFlightMission’: [],
‘CitationSubset’: [‘IM’],
‘GeneralNote’: [],
‘KeywordList’: [],
‘PMID’: StringElement(‘16904174’, attributes={‘Version’: ‘1’}),
‘DateCompleted’: {‘Year’: ‘2006’, ‘Month’: ’10’, ‘Day’: ’31’},
‘DateRevised’: {‘Year’: ‘2023’, ‘Month’: ’02’, ‘Day’: ’07’},
‘Article’: DictElement({‘ELocationID’: [], ‘ArticleDate’: [DictElement({‘Year’: ‘2006’, ‘Month’: ’08’, ‘Day’: ’10’},
・
・
・
と大量にそのIDの論文の情報が入っています。
私達に馴染みのある情報は
article[‘PubmedArticle’][0][‘MedlineCitation’][‘Article’]
の項目に入っており、内容を表示すると
print(article['PubmedArticle'][0]['MedlineCitation']['Article'])
DictElement({
‘ELocationID’: [],
‘ArticleDate’: [DictElement({‘Year’: ‘2006’, ‘Month’: ’08’, ‘Day’: ’10’}, attributes={‘DateType’: ‘Electronic’})],
‘Language’: [‘eng’],
‘Journal’: {‘ISSN’: StringElement(‘0092-8674’, attributes={‘IssnType’: ‘Print’}), ‘JournalIssue’: DictElement({‘Volume’: ‘126’, ‘Issue’: ‘4’, ‘PubDate’: {‘Year’: ‘2006’, ‘Month’: ‘Aug’, ‘Day’: ’25’}}, attributes={‘CitedMedium’: ‘Print’}),
‘Title’: ‘Cell’, ‘ISOAbbreviation’: ‘Cell’},
‘ArticleTitle’: ‘Induction of pluripotent stem cells from mouse embryonic and adult fibroblast cultures by defined factors.’,
‘Pagination’: {‘StartPage’: ‘663’, ‘EndPage’: ‘676’, ‘MedlinePgn’: ‘663-76’},
‘Abstract’: {‘AbstractText’: [‘Differentiated cells can be reprogrammed to an embryonic-like state by transfer of nuclear contents into oocytes or by fusion with embryonic stem (ES) cells. ・・・’]},
‘AuthorList’:・・・),
‘PublicationTypeList’: [StringElement(‘Journal Article’, attributes={‘UI’: ‘D016428’}), StringElement(“Research Support, Non-U.S. Gov’t”, attributes={‘UI’: ‘D013485’})]}, attributes={‘PubModel’: ‘Print-Electronic’})
のように、
雑誌名、ページ数、言語、作者情報、アブストラクト(抄録)、タイトル、筆者、投稿の種類(Journal ArticleやReviewなど)の情報が辞書型で表示されます。
そこからアブストラクトの情報は
abstract = article['MedlineCitation']['Article']['Abstract']['AbstractText'][0]で入手することができます。
実際の構造をみると面白いです。
このように情報が入手できれば、
「自分のほしいIDのリストを手に入れる」
↓
「そのIDに関連付けた情報を手に入れて一覧にする」
という流れを、pythonで簡単に行うことが出来るようになります。
Pythonのように、テキストの扱いが簡単だと、直感的にもわかりやすいですね。
一度やってみましょう。
from Bio import Entrez
Entrez.email = "使う人のEmailアドレス"
def search_articles(keyword, max_results=10):
handle = Entrez.esearch(db='pubmed',
sort='relevance',
retmax=str(max_results),
term=keyword)
results = Entrez.read(handle)
return results
def fetch_abstract(pmid):
handle = Entrez.efetch(db='pubmed', id=pmid, retmode='xml')
article = Entrez.read(handle)['PubmedArticle'][0]
abstract = article['MedlineCitation']['Article']['Abstract']['AbstractText'][0]
return abstract