ここではDICOMデータをPythonのpydicomというツールを用いて開きます。
さきにコードを書いておくと
import pydicom
from matplotlib import pyplot as plt
ds = pydicom.read_file('test.dcm')
plt.imshow(ds.pixel_array, cmap='gray')
plt.show()こんな感じです。
必要なライブラリ(Pythonのツール)は
- matplotlib
- pydicom
です。
解説を加えていきます。
1.pydicomモジュールをインストールして使用する
Pythonと画像データは相性がいいです。
なぜかというと、画像は縦、横の白黒データの羅列で表せます。
行列の扱いはPythonの拡張モジュールのnumpyの得意とするところであり、
行列の描画はmatplotlibでできるからです。
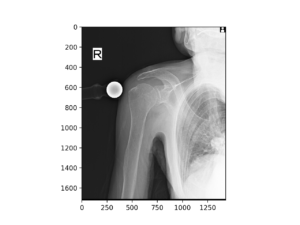
早速レントゲン画像を見てみます。
Macを使用した例です。
Google Colabolatoryでも同じ作業でできます。
numpy, matplotlibはanacondaでインストールしておきます。
まず、「pydicom」というモジュールをインストールします。
例として肩のレントゲンを読み込みます。
pythonやipythonの対話モード上で行います。
まずpydicomとmatplotlibのpyplotをインポートします。
from matplotlib import pyplot as plt
つぎにDICOMデータを読み込み、dsという変数に入れます。
ここでは練習用の肩のレントゲンデータ 「test.dcm」 というファイルを使うとします。
ds = pydicom.read_file(‘test.dcm’)
print(ds)
(0008, 0008) Image Type CS: [‘ORIGINAL’, ‘PRIMARY’]
(0008, 0016) SOP Class UID UI: Computed Radiography Image Storage
(0008, 0018) SOP Instance UID UI: DICOMのidが表示される
(0008, 0020) Study Date DA: ’20xx0xxx’ 日付が表示される
(0008, 0023) Content Date DA: ’20xx0xxx’ 日付が表示される
(0008, 0030) Study Time TM: ‘170753.0’ 撮影時刻
(0008, 0033) Content Time TM: ‘170956.0’
(0008, 0050) Accession Number SH: ‘xxxxx8100026727’ 撮影 id
(0008, 0060) Modality CS: ‘CR’ レントゲン
(0008, 0070) Manufacturer LO: ‘KONICA MINOLTA’など
(0008, 0080) Institution Name LO: [‘施設名’]
/
/
数十行続く
DICOMのラベルがでます。
dsはpydicomのデータセットです。typeで形式を見ることができます。
2.データフレームのpixel_array属性で画像の濃淡を取得
このデータセットのなかで
ds.pixel_array
に画像の濃度の情報が格納されています。
[478, 482, 478, …, 63, 63, 0],
[468, 473, 478, …, 63, 63, 0],
…,
[ 0, 0, 0, …, 0, 0, 0],
[ 0, 0, 0, …, 0, 0, 0],
[ 0, 0, 0, …, 0, 0, 0]], dtype=uint16)
濃淡が行列で入っています。
縦1722ピクセル、横1430ピクセルです
3.Matplotlibで濃淡のデータを表示する
この行列をmatplotlibで白黒表示すればレントゲンがでます。
# グレースケールのためのcmap=”gray”
plt.show()
レントゲン画像が表示されます。
4.まとめ
まとめると
「test.dcm」というDICOMファイルを表示するには
pipでpydicomをインストールしてから
import pydicom
from matplotlib import pyplot as plt
ds = pydicom.read_file('test.dcm')
plt.imshow(ds.pixel_array, cmap='gray')
plt.show()となります。
結構シンプル。