このページは表面データ構築ソフト 3D Slicerの使用方法の2ページ目です。
3.ビューアーの設定
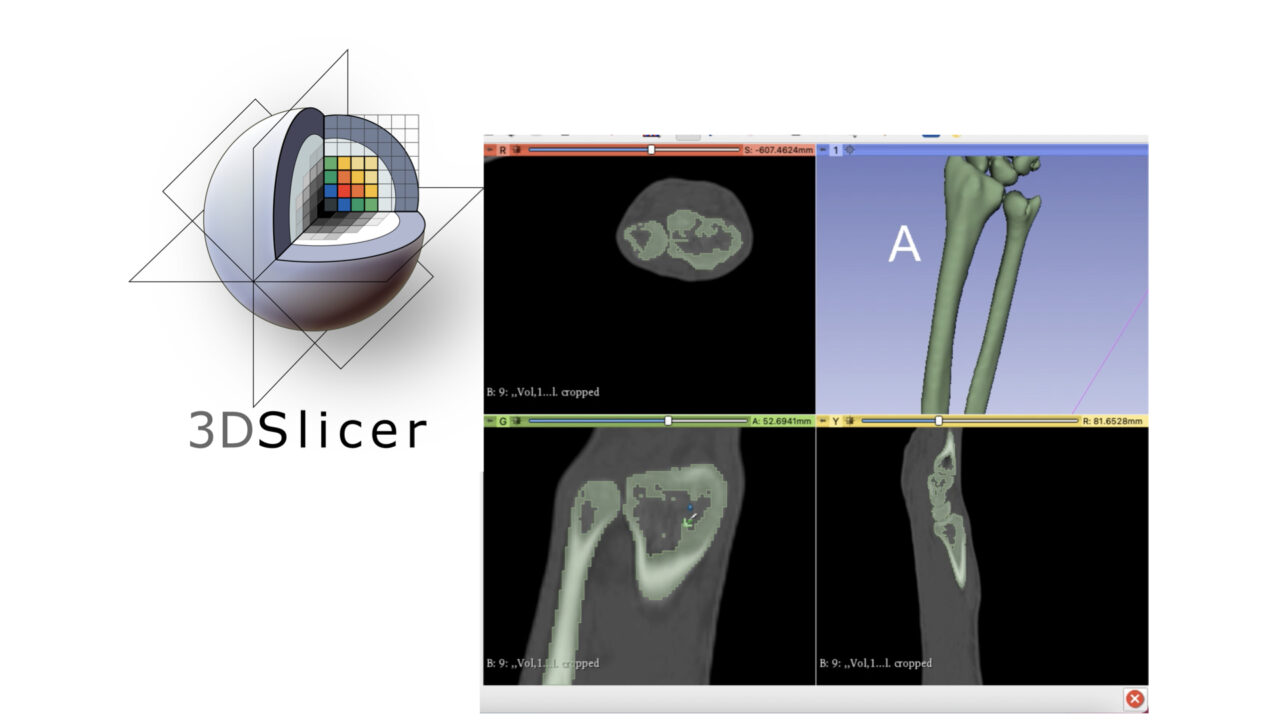
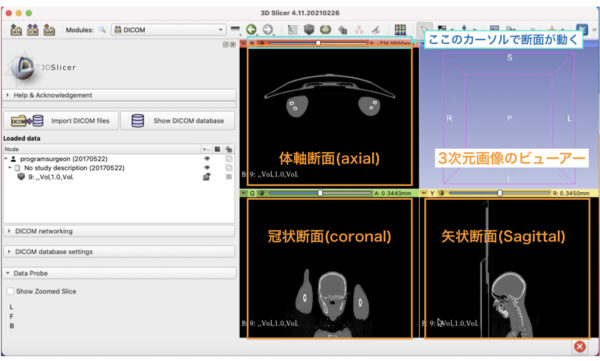
初期の画面は4分割されています。
このうち3つは、3方向のスライスです。一般的にはAxial(体軸断面), Sagittal(矢状断面), Coronal(冠状断面)を表します。
ひとつ青い画面が表示されていますが、ここには各スライスでマーキングされた位置情報が3次元表面画像として表示されます。
まずは画像の濃淡など見え方を調整します。
上のプルダウンメニューから、Volumesという項目を開きます。
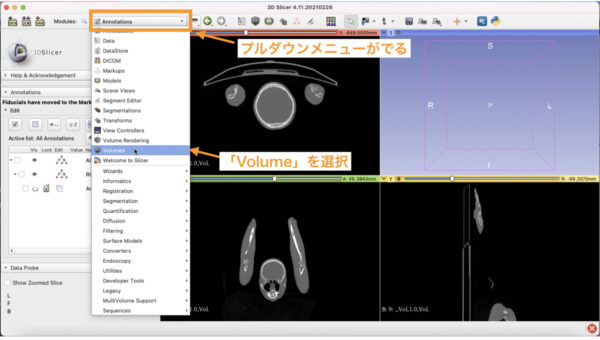
まんなかあたりに、Viewerの色の条件を決めるスケールがあります。
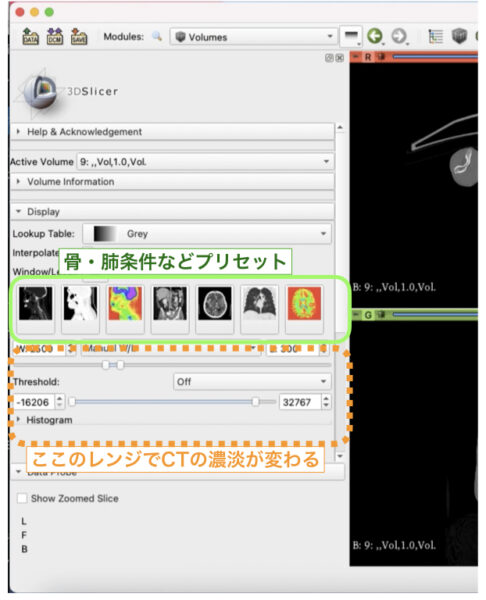
ここでスケールの値を変えると、ビューアー濃淡の範囲を変更して、画像の見え方を変える事ができます。
真ん中あたりにある画像の絵を選ぶと、骨の条件や肺の条件などプリセットを選ぶことができます。
まずは自分の関心領域がよく観察できるよう設定しておきます。
4.関心領域のデータを切り取る
①領域の選択(Volume Rendering)
全身CTなどから骨一つを取り出す時など、CTの量が膨大だと操作一つ一つに解析時間が長くかかります。
そのため、焦点を絞ってCT画像を切り出し、切り出した画像で作業をすることでパソコンにかかる負担をへらすことができます。
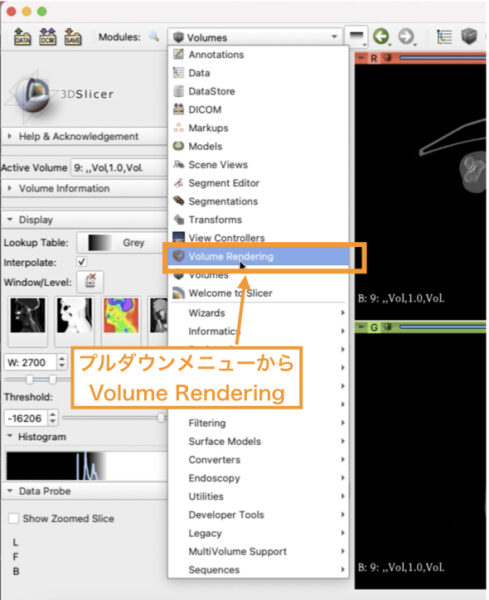
まずプルダウンメニューから
「Volume Rendering」
を選びます。
Display ROIという目のマークをチェックすると、目が開き、関心領域のボックスが現れます。
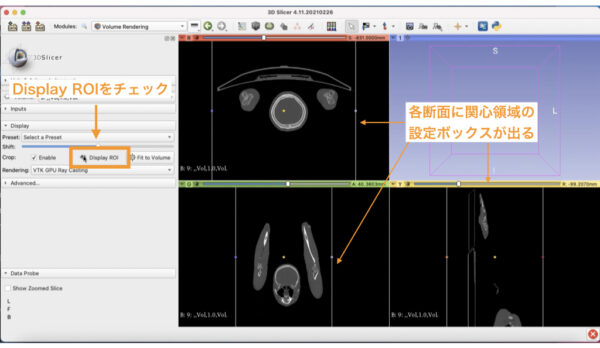
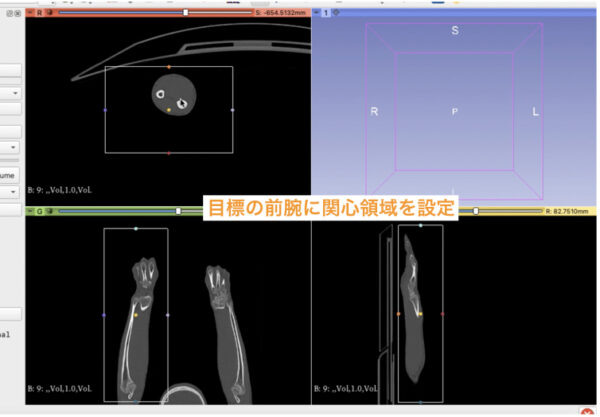
このボックスをすべてのスライスで調節して、自分の関心領域(今回は前腕)が入るように設定します。
データ負担をへらす目的なので、関心領域が切れてしまわない範囲でざっくりでよいです。
② 関心領域の切り出し(Crop Volume)
関心領域が選択できたら、切り取りを行います。
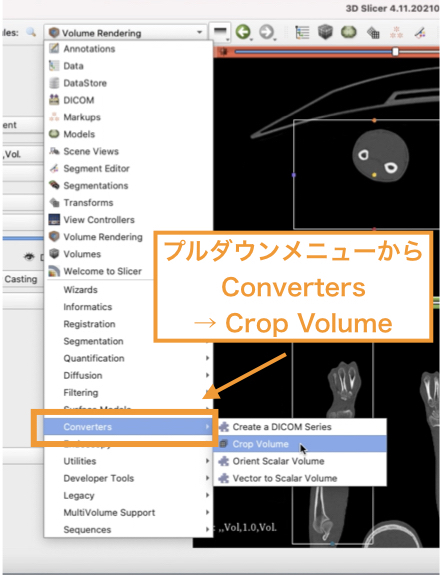
プルダウンメニューから、
「Converters」 → 「Crop Volume」
を選びます。
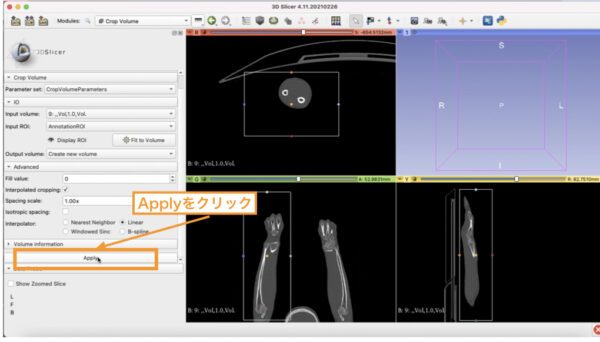
Crop Volumeのメニューで「Apply」をクリックすると
データが切り取られます。
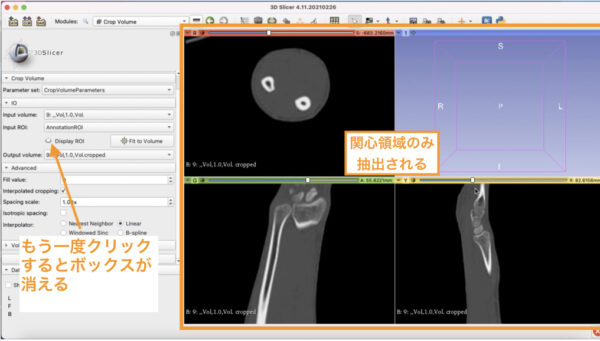
もう一度Display ROIの目をクリックすると関心領域のボックスが消えます。
このさきの編集では邪魔になるので消しておきましょう。
5.骨のセグメンテーションと3D表示
① セグメンテーションツールとセグメントの設定
橈骨の表面データを作るのが目的なので、橈骨の骨を色分けします。
この作業をセグメンテーション(Segmentation)といいます。
SlicerではSegmentation Editorというメニューでたくさんのツールが使えます。
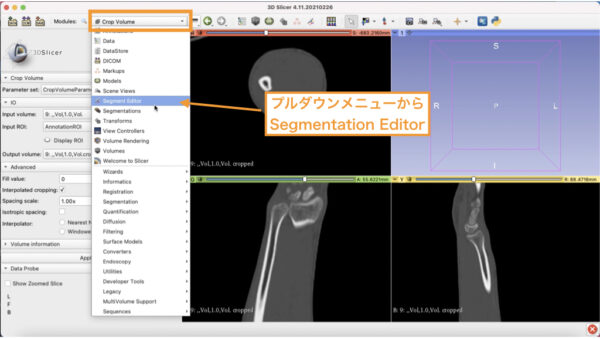
ここで、作業を行う対象の画像が、切り出されたものであることを確認しましょう。
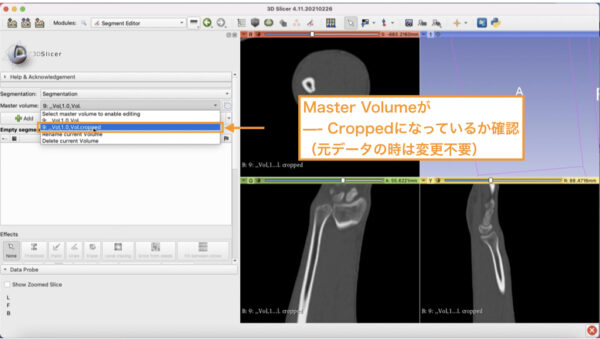
Master Volumeという項目で、
—Croppedというボリュームデータが選ばれていればOKです。
「Add」ボタンでセグメントを追加します。
ここでは名前を「radius」(橈骨)としておきます。

② 骨領域の抽出
CTの値から骨の領域を抽出します。
セグメンテーションツールから、Threshold(閾値)ボタンをえらびます。
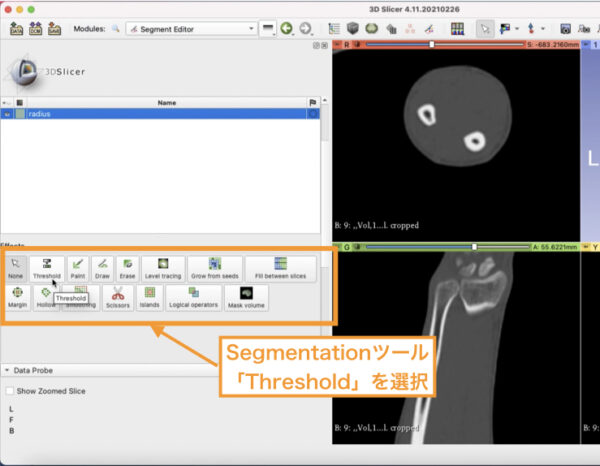
これは、指定範囲の領域にあてはまるCT値をもった部分だけを色塗りするツールです。
骨は大体250から300以上のCT値になるため、ここでは下限を270に設定します。
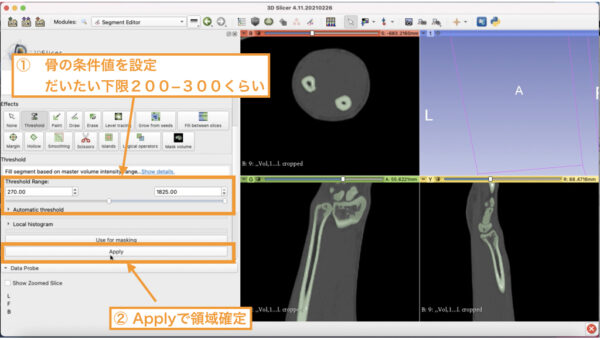
骨に当たるすべての領域に色が付きます。
「Apply」を押すと、色の点滅がとまり、
セグメント「radius」に選択領域が加わります。
③ 3D画像の表示
塗られた領域を表面画像に変換してみます。
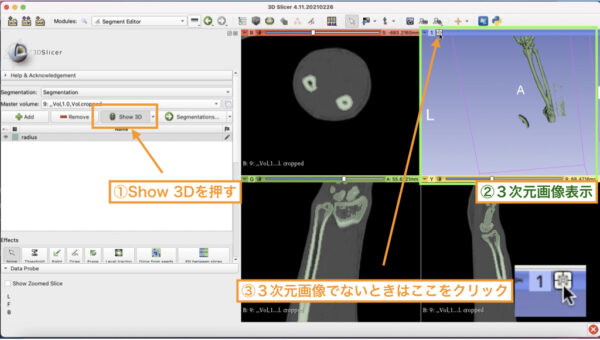
「Show 3D」というボタンをクリックすると、
塗られているすべてのセグメントが3次元画像化されます。
3次元画像は右上に表示されます。
この時、何も表示されない場合は、3次元画像ウィンドウの上の小さい
十字座標のボタンをクリックしてみましょう。
位置合わせをおこなってくれて、表面データの中心に視野が設定され、表面モデルが観察できるようになります。